Systembiologische Analysen mit dem Moos Physcomitrella patens
Einleitung
In Zeiten des „next-generation-sequencing“ sind komperative genomische Analysen nicht mehr auf eine geringe Anzahl an Spezies beschränkt, da quasi monatlich neu sequenzierte Genome veröffentlicht werden. Stattdessen ermöglichen frei verfügbare Sequenzdatensätze die evolutionäre Analyse von verschiedensten Spezies in einem Speziesbereich, der sowohl nah als auch fernverwandte Arten mit einschließt.
Methoden
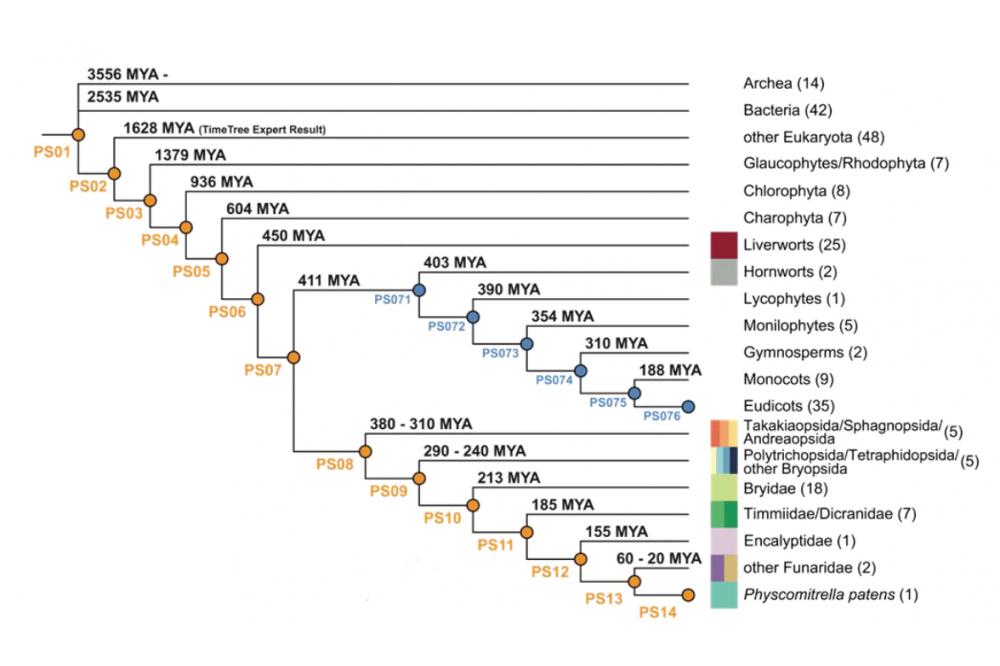
Im Zuge des Projekts „Systembiologische Analysen mit dem Moos Physcomitrella patens“ wurden sowohl Genom- als auch Transkriptomdaten verwendet, um den phylogenetischen Ursprung von Physcomitrella patens Genen herauszufinden. Neben frei verfügbaren Datensätzen lagen uns noch nicht veröffentlichte Daten des „One thousands plants (1KP) Projects“ (www.onekp.com) vor, um die Speziesdichte innerhalb der Moose zu erhöhen und zusätzlich Genomduplikationen innerhalb der Moose zu bestimmen. Das Genalter einzelner Physcomitrella patens Gene wurde mit der Hilfe von einem vorhandenen phylogenetischem Baum [1] und phylostratigraphischen Methoden [2] bestimmt. Hierzu wird zunächst eine individuelle phylostartigraphische Karte von Physcomitrella patens erstellt (Abb. 1). Nach rechenintensiven Sequenzvergleichen3 werden die Gene mittels eigens entwickelter python Skripte in einzelne Phylostrata (PS) einsortiert und erhalten damit einen Zeitstempel. Dieser Zeitstempel bzw. das Genalter kann dann genutzt werden, um Genexpressionswerte oder andere Merkmale der Gene zu korrelieren, wie es an anderer Stelle schon gezeigt werden konnte [4]. Zusätzlich können die Ergebnisse auch helfen die Evolution von einzelnen Genfamilien der Moose zu entschlüsseln. Für diese Analysen werden Seqeunzabgleiche mit Genpositionen auf den einzelnen Chromosomen zusammengeführt, um zunächst synthenische Bereiche zu finden. Diese Information über orthologe Proteine kann dann wiederum mit dem Genalter korreliert und in chromosomweiten Abbildungen visualisiert werden (circos.ca).
Ergebnisse
Die Analyse konnte zeigen, dass innerhalb von Physcomitrella patens eine Vielzahl speziesspezifischer Gene „orphan genes“ vorliegen, die nicht in anderen Moosen vorhanden sind. Für einen Teil dieser „orphan genes“ konnten andere Forschergruppen bereits eine Funktion entschlüsseln5, für andere Gene im Organismus steht diese Aufgabe noch aus und muss in weiteren Studien geklärt werden. Die Analyse der Genomduplikationen innerhalb der Moose deutet auf einen Anreicherung von Duplikationsevents vor 70 Millionen Jahren hin, wie sie aus anderen Veröffentlichungen schon vorhergesagt wurde6. Dieser phylostratigraphische Ansatz ist natürlich nicht auf das Moos Physcomitrella patens beschränkt und wird in Kooperation mit Ivo Grosse von der Martin-Luther Universität Halle (Saale) in einem kritischen Licht beleuchtet, um die Gruppierung in die einzelnen PS zu optimieren und biologisch zu verifizieren. Ziel ist es anhand der gefundenen Ergebnisse Schlüsselereignisse innerhalb der Evolution von Landpflanzen zu detektieren und mit dem ersten Erscheinen von Genfamilien innerhalb bestimmter Spezies in Einklang zu bringen.